AbdomenAtlas 3.0|医学影像分析数据集|肿瘤检测数据集
收藏arXiv2025-01-09 更新2025-01-10 收录
下载链接:
https://github.com/MrGiovanni/RadGPT
下载链接
链接失效反馈资源简介:
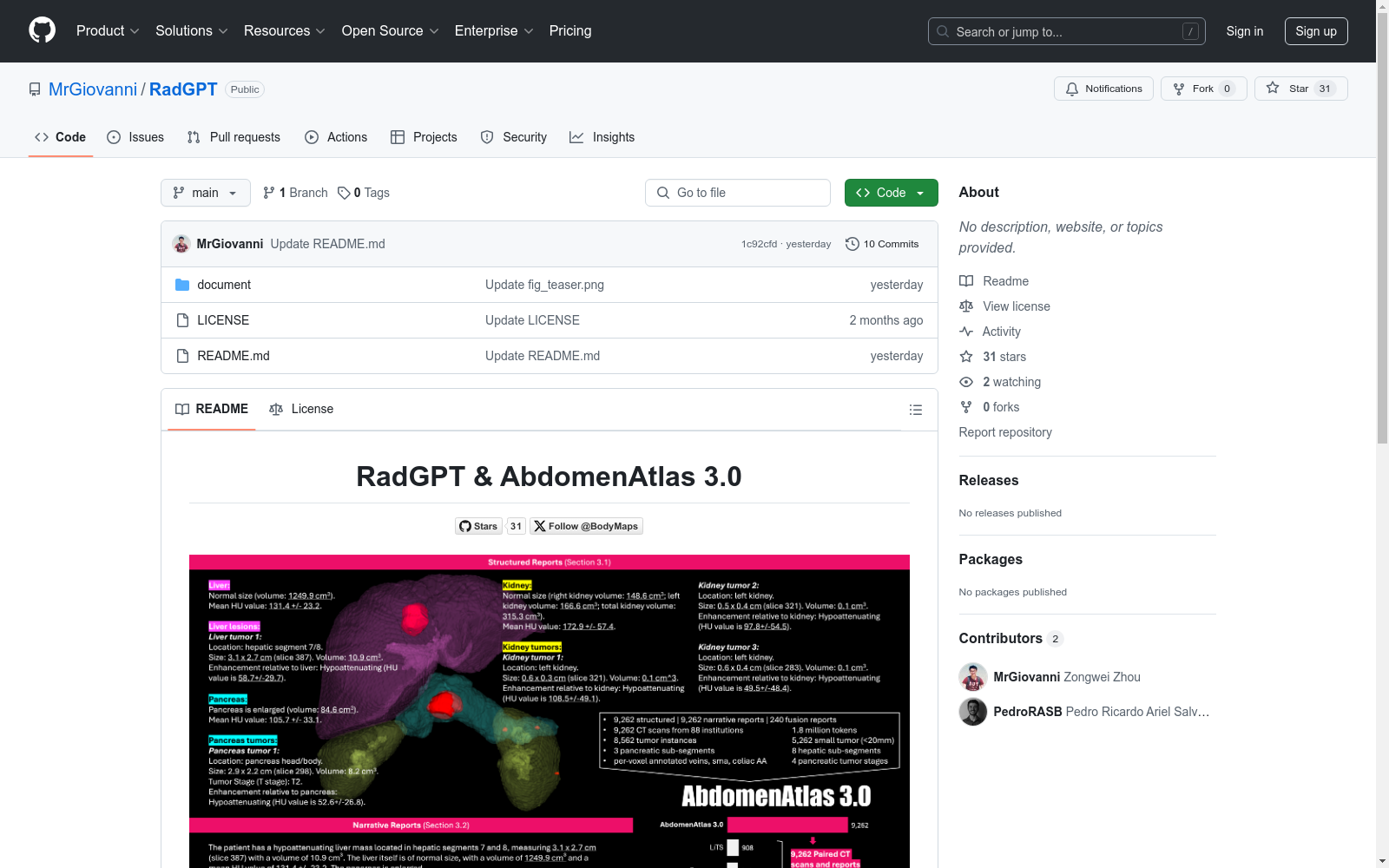
AbdomenAtlas 3.0是一个大规模的图像-文本肿瘤数据集,由约翰霍普金斯大学等机构创建,包含9262个3D CT扫描,每个扫描都有详细的肿瘤注释和报告。数据集涵盖了3036个肝脏肿瘤、354个胰腺肿瘤和4239个肾脏肿瘤,其中5262个为小肿瘤(≤2cm)。数据集还提供了胰腺癌分期(T1-T4)的详细注释,以及肝脏和胰腺子段的体素级分割。该数据集旨在通过自动化报告生成提高肿瘤检测的准确性和效率,特别是在早期癌症检测方面。
提供机构:
约翰霍普金斯大学, 博洛尼亚大学, 意大利理工学院, 加州大学旧金山分校, 伊利诺伊大学厄巴纳-香槟分校, 洛桑联邦理工学院
创建时间:
2025-01-09
AI搜集汇总
数据集介绍
构建方式
AbdomenAtlas 3.0的构建基于大规模的3D CT扫描数据,涵盖了9,262个CT扫描,每个扫描均包含逐体素的肿瘤注释和详细的放射学报告。数据集的构建过程首先通过RadGPT模型对肿瘤及其周围解剖结构进行分割,随后将这些信息转化为结构化和叙述性报告。这些报告不仅提供了肿瘤的大小、形状、位置、衰减和体积等详细信息,还描述了肿瘤与周围血管和器官的相互作用。为确保报告的准确性,数据集经过放射科医生的审查和修订,最终形成了包含1,843,262个文本标记和2,789,975张图像的公开数据集。
特点
AbdomenAtlas 3.0的特点在于其大规模、高精度的3D CT扫描数据,涵盖了肝脏、胰腺和肾脏的多种肿瘤类型。数据集特别关注小肿瘤(≤2 cm)的检测,提供了3,036个肝脏肿瘤、354个胰腺肿瘤和4,239个肾脏肿瘤的详细注释。此外,数据集还首次公开了胰腺癌分期(T1-T4)的详细注释,以及肝脏和胰腺亚段的逐体素分割信息。这些特点使得AbdomenAtlas 3.0成为首个公开的包含胰腺癌分期和胰腺亚段分割的医学图像数据集。
使用方法
AbdomenAtlas 3.0的使用方法主要包括通过RadGPT模型生成结构化和叙述性报告。用户可以利用数据集中的逐体素注释和CT扫描数据,训练和评估AI模型在肿瘤检测和报告生成方面的性能。数据集还可用于研究肿瘤的早期检测、分期和与周围解剖结构的相互作用。此外,AbdomenAtlas 3.0提供了240份人机融合报告,结合了结构化报告和临床笔记,进一步扩展了数据集的应用场景。用户可以通过访问GitHub上的代码和模型,利用该数据集进行自动化报告生成和肿瘤分析。
背景与挑战
背景概述
AbdomenAtlas 3.0 是一个大规模的三维腹部CT扫描图像-文本肿瘤数据集,由约翰霍普金斯大学、博洛尼亚大学、加州大学旧金山分校等机构的研究团队于2025年发布。该数据集包含9,262个CT扫描,每个扫描均带有逐体素的肿瘤标注和详细的放射学报告,涵盖了肝脏、胰腺和肾脏的肿瘤信息。AbdomenAtlas 3.0 的创建旨在解决放射科医生在生成肿瘤相关报告时面临的高工作量和高错误率问题,特别是在小肿瘤(≤2厘米)的检测和报告生成方面。该数据集不仅提供了肿瘤的大小、形状、位置等详细信息,还首次公开了胰腺癌分期(T1-T4)和胰腺亚段(头、体、尾)的逐体素标注,为腹部CT图像的自动化报告生成提供了重要的研究基础。
当前挑战
AbdomenAtlas 3.0 面临的挑战主要集中在两个方面。首先,小肿瘤的检测和报告生成是当前领域的主要难题,尤其是在胰腺和肝脏等复杂解剖结构中,小肿瘤的识别和定位对放射科医生和AI模型都提出了极高的要求。尽管AbdomenAtlas 3.0 提供了大量小肿瘤的标注,但其检测的敏感性和特异性仍需进一步提升。其次,数据集的构建过程中,逐体素标注的生成和验证是一个耗时且复杂的过程,尤其是在胰腺和肝脏的亚段分割中,如何确保标注的精确性和一致性是一个技术挑战。此外,如何将结构化报告转化为符合不同医院风格的叙述性报告,同时保持医学信息的准确性,也是数据集应用中的一大挑战。
常用场景
经典使用场景
AbdomenAtlas 3.0数据集在医学影像分析领域具有广泛的应用,尤其是在腹部CT扫描的肿瘤检测与报告生成中。该数据集通过提供大量的三维CT扫描图像及其对应的肿瘤标注和报告,为研究人员和临床医生提供了丰富的资源。其经典使用场景包括自动化肿瘤检测、肿瘤分期、以及生成结构化和叙事性报告。特别是在肝脏、胰腺和肾脏肿瘤的早期检测中,AbdomenAtlas 3.0通过其详细的体素级标注,显著提升了模型的检测精度。
实际应用
在实际应用中,AbdomenAtlas 3.0为临床医生提供了强大的工具,帮助他们更快速、准确地生成肿瘤报告。通过RadGPT模型,医生可以自动化地生成结构化和叙事性报告,减少了人工编写报告的时间和错误率。此外,该数据集在胰腺癌、肝癌和肾癌的早期诊断中具有重要应用价值,尤其是在小肿瘤的检测和分期方面。AbdomenAtlas 3.0还为AI模型的训练和评估提供了标准化的基准,推动了医学影像AI技术的实际落地。
衍生相关工作
AbdomenAtlas 3.0的发布催生了一系列相关研究工作,尤其是在腹部CT影像的自动化报告生成和肿瘤检测领域。基于该数据集,研究人员开发了多种先进的AI模型,如RadGPT,这些模型在肿瘤检测、分期和报告生成方面表现出色。此外,AbdomenAtlas 3.0还为其他研究提供了数据支持,推动了肝脏、胰腺和肾脏肿瘤的自动化分析技术的发展。该数据集的广泛应用也促进了医学影像AI模型的标准化评估,为未来的研究奠定了坚实的基础。
以上内容由AI搜集并总结生成



