ChemBL|化学信息学数据集|药物发现数据集
收藏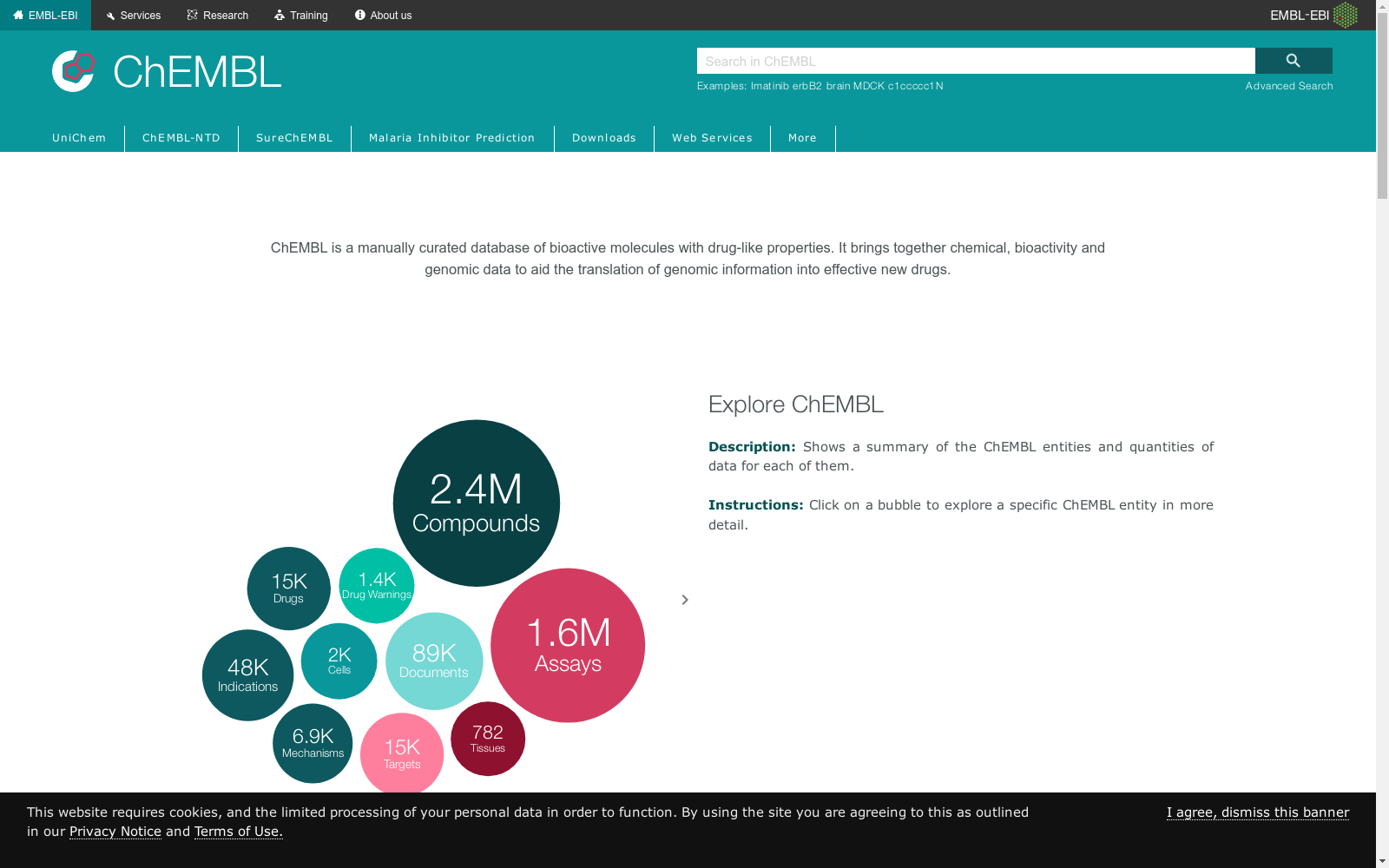
- ChemBL项目由欧洲生物信息学研究所(EBI)启动,旨在创建一个全面的化学数据库,用于药物发现和化学信息学研究。
- ChemBL数据库首次公开发布,提供了一个包含大量化学实体和生物活性数据的资源,为学术界和工业界的研究人员提供了宝贵的工具。
- ChemBL数据库进行了重大升级,引入了更多的化学和生物数据,增强了其作为药物发现平台的功能。
- ChemBL数据库被整合到ChEMBLdb中,这是一个更为先进和用户友好的平台,提供了更强大的搜索和分析工具。
- ChEMBLdb发布了第17版,引入了新的数据类型和改进的数据处理算法,进一步提升了其作为化学信息学工具的价值。
- ChEMBLdb发布了第24版,增加了对大规模数据集的支持,并引入了机器学习算法,以提高数据分析的准确性和效率。
- ChEMBLdb发布了第30版,继续扩展其数据覆盖范围,并引入了新的数据可视化工具,以支持更复杂的药物发现研究。
LFW
人脸数据集;LFW数据集共有13233张人脸图像,每张图像均给出对应的人名,共有5749人,且绝大部分人仅有一张图片。每张图片的尺寸为250X250,绝大部分为彩色图像,但也存在少许黑白人脸图片。 URL: http://vis-www.cs.umass.edu/lfw/index.html#download
AI_Studio 收录
rag-datasets/rag-mini-bioasq
该数据集主要用于问答和句子相似性任务,涉及生物医学领域。数据集包含两个配置:text-corpus和question-answer-passages,分别对应不同的数据文件路径。数据集来源于BioASQ任务11b的训练数据集,并通过`generate.py`脚本生成了子集。
hugging_face 收录
Med-MAT
Med-MAT是一个包含106个开源医学数据集的视觉问答(VQA)数据集,旨在推动医学多模态大语言模型(MLLMs)的泛化实验和训练。数据集通过将图像-标签对转换为VQA格式,展示了组合泛化(CG)是MLLMs理解未见图像的关键机制。数据集包括106个医学数据集的问答对、53个按模态、解剖区域和任务(MAT)分类的子集的问答对,以及部分数据集的图像下载链接。
huggingface 收录
PQAref
PQAref数据集是一个用于生物医学领域参考问答任务的数据集,旨在微调大型语言模型。该数据集包含三个部分:指令(问题)、摘要(从PubMed检索的相关摘要,包含PubMed ID、摘要标题和内容)和答案(预期答案,包含PubMed ID形式的参考)。数据集通过半自动方式创建,利用了PubMedQA数据集中的问题。
huggingface 收录
LIDC-IDRI
LIDC-IDRI 数据集包含来自四位经验丰富的胸部放射科医师的病变注释。 LIDC-IDRI 包含来自 1010 名肺部患者的 1018 份低剂量肺部 CT。
OpenDataLab 收录
