lhallee/Full_PDB_Contacts|生物信息学数据集|蛋白质结构分析数据集
收藏数据集概述
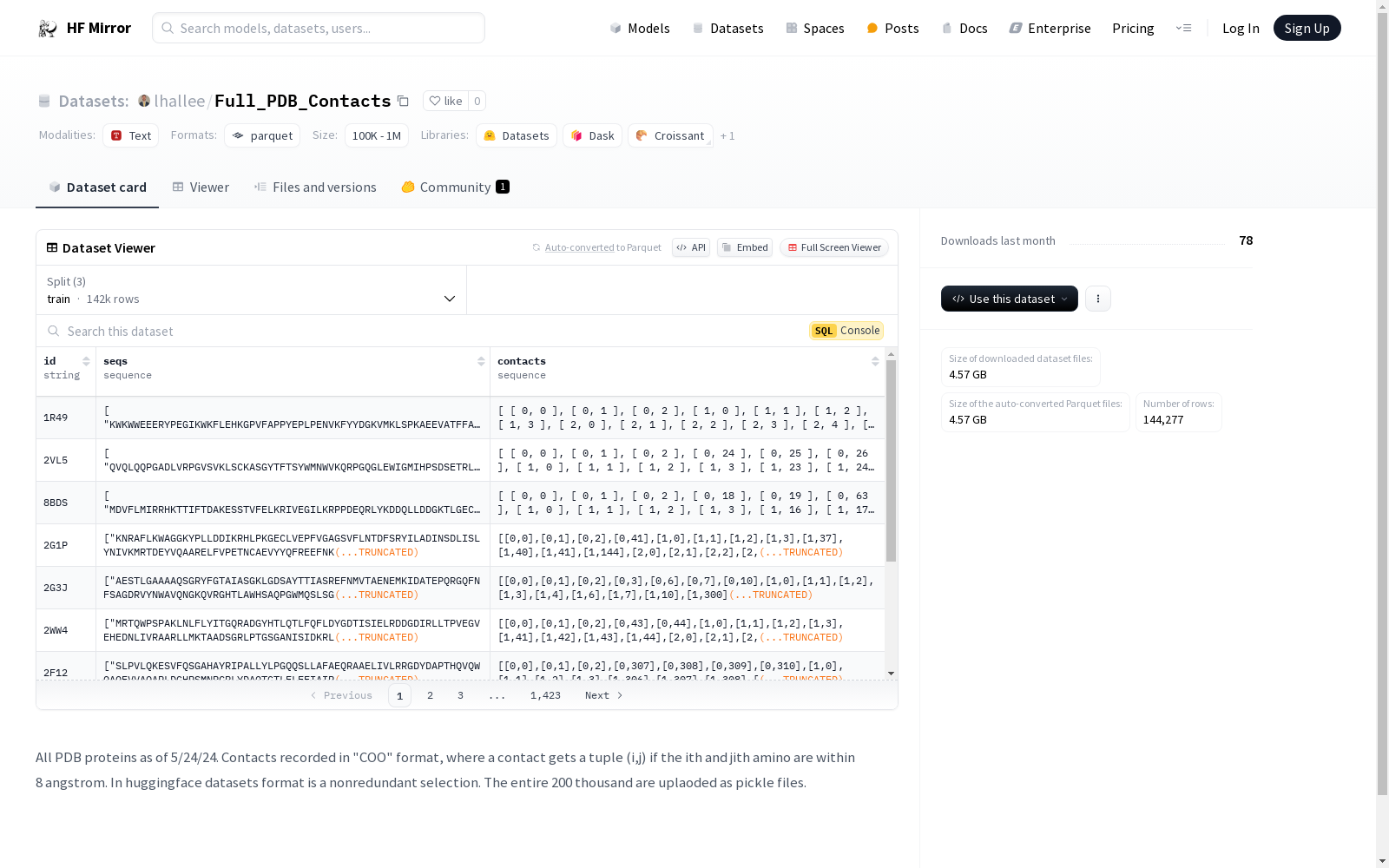
数据集特征
- id: 数据类型为字符串。
- seqs: 数据类型为字符串,具有序列属性。
- contacts: 数据类型为无符号16位整数,具有序列属性。
数据集分割
- train: 包含90,193个样本,总大小为3,896,443,787字节。
数据集大小
- 下载大小: 1,539,844,300字节。
- 数据集大小: 3,896,443,787字节。
配置
- config_name: default
- data_files:
- split: train
- path: data/train-*
WLASL, MSASL, NMFs-CSL, SLR500, Slovo, BOBSL, 27 Class Sign Language Dataset, AUTSL, BosphorusSign22k, GSL, LSA16, LSA64, Rendered Handpose Dataset, YouTube-ASL, LSFB-ISOL, ASLLVD, AASL, KArSL, BdSLImset, HaGRID, Phoenix-2014, Phoenix-2014T
该仓库收集了多种与手语识别和翻译相关的数据集,旨在为研究者、开发者和爱好者提供一个集中的资源。数据集包括不同类型(如RGB、深度、骨骼)和来自不同国家的数据,用于支持手语识别和翻译技术的研究。
github 收录
Med-MAT
Med-MAT是一个包含106个开源医学数据集的视觉问答(VQA)数据集,旨在推动医学多模态大语言模型(MLLMs)的泛化实验和训练。数据集通过将图像-标签对转换为VQA格式,展示了组合泛化(CG)是MLLMs理解未见图像的关键机制。数据集包括106个医学数据集的问答对、53个按模态、解剖区域和任务(MAT)分类的子集的问答对,以及部分数据集的图像下载链接。
huggingface 收录
红外谱图数据库
收集整理红外谱图实验手册等数据,建成了红外谱图数据库。本数据库收录了常见化合物的红外谱图。主要包括化合物数据和对应的红外谱图数据。其中,原始红外谱图都进行了数字化处理,从而使谱峰检索成为可能。用户可以在数据库中检索指定化合物的谱图,也可以提交谱图/谱峰数据,以检索与之相似的谱图数据,以协助进行谱图鉴定。
国家基础学科公共科学数据中心 收录
HazyDet
HazyDet是由解放军工程大学等机构创建的一个大规模数据集,专门用于雾霾场景下的无人机视角物体检测。该数据集包含383,000个真实世界实例,收集自自然雾霾环境和正常场景中人工添加的雾霾效果,以模拟恶劣天气条件。数据集的创建过程结合了深度估计和大气散射模型,确保了数据的真实性和多样性。HazyDet主要应用于无人机在恶劣天气条件下的物体检测,旨在提高无人机在复杂环境中的感知能力。
arXiv 收录
MinneApple 苹果检测数据集
MinneApple 是一个用于苹果检测和分割的基准数据集。该数据集使用多边形掩码为每个对象实例进行标注,以帮助进行精确的对象检测、定位、和分割。此外,该数据集提供了基于补丁的聚类水果计数数据。该数据集在 1000 张图像中包含超过 41,0000 个带标注的对象实例。
超神经 收录
