enzyme_kcat|酶催化效率数据集|生物信息学数据集
收藏数据集概述
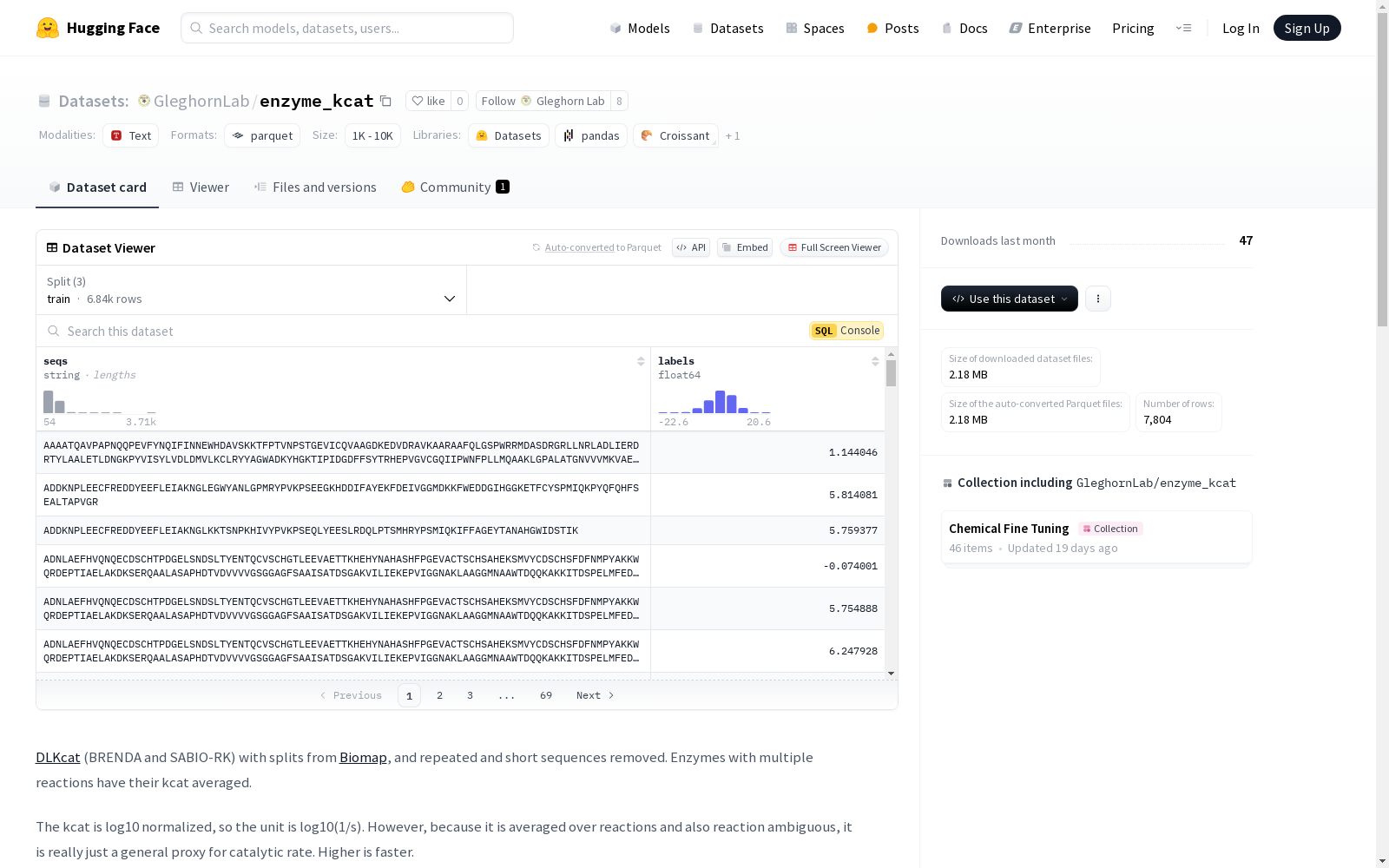
数据集信息
- 特征:
seqs: 序列数据,类型为字符串 (string)labels: 标签数据,类型为浮点数 (float64)
- 分割:
train: 训练集,包含6837个样本,占用2933951字节valid: 验证集,包含498个样本,占用217038字节test: 测试集,包含469个样本,占用204262字节
- 下载大小: 2178499字节
- 数据集总大小: 3355251字节
配置
- 配置名称:
default- 数据文件路径:
train:data/train-*valid:data/valid-*test:data/test-*
- 数据文件路径:
数据处理
- 数据来源:
- kcat值:
- kcat值经过log10归一化处理,单位为log10(1/s)。
- 由于kcat值在反应中被平均化且存在反应模糊性,因此它只是一个催化速率的通用代理,数值越高表示催化速率越快。
数据处理代码
python import pandas as pd from datasets import Dataset, DatasetDict, concatenate_datasets
def process_dataset(dataset_dict): precedence = [train, valid, test] # 为每个数据集添加split列 for split in dataset_dict.keys(): dataset_dict[split] = dataset_dict[split].add_column(split, [split]*len(dataset_dict[split])) # 将所有分割合并为一个数据集 all_data = concatenate_datasets([dataset_dict[split] for split in dataset_dict.keys()]) # 转换为pandas DataFrame df = all_data.to_pandas() # 移除长度小于50的序列 df[seq_length] = df[seqs].apply(len) df = df[df[seq_length] >= 50] # 按seqs分组以查找重复项并平均标签 def aggregate_group(group): avg_label = group[labels].mean() # 将序列分配到其出现的最高优先级分割 for p in precedence: if p in group[split].values: selected_split = p break return pd.Series({labels: avg_label, split: selected_split}) df_grouped = df.groupby(seqs).apply(aggregate_group).reset_index() # 将DataFrame拆分为原始分割,且不包含重叠序列 new_dataset_dict = DatasetDict() for split in precedence: df_split = df_grouped[df_grouped[split] == split] new_dataset_dict[split] = Dataset.from_pandas(df_split[[seqs, labels]], preserve_index=False) return new_dataset_dict
IST-3 CT Head Scans
IST-3 CT头部扫描数据集由爱丁堡大学临床脑科学中心创建,包含10,659个CT系列,用于研究颅内动脉钙化的分割。数据集来源于第三届国际中风试验(IST-3),涉及3035名急性缺血性中风患者的非增强CT扫描。数据集创建过程中,通过与模板配准和质量控制,确保了数据的有效性和准确性。该数据集主要用于支持深度学习方法在中风风险评估中的应用,特别是在颅内动脉钙化的自动量化方面。
arXiv 收录
Yahoo Finance
Dataset About finance related to stock market
kaggle 收录
HotpotQA
HotpotQA 是收集在英语维基百科上的问答数据集,包含大约 113K 众包问题,这些问题的构建需要两篇维基百科文章的介绍段落才能回答。数据集中的每个问题都带有两个黄金段落,以及这些段落中的句子列表,众包工作人员认为这些句子是回答问题所必需的支持事实。 HotpotQA 提供了多种推理策略,包括涉及问题中缺失实体的问题、交叉问题(什么满足属性 A 和属性 B?)和比较问题,其中两个实体通过一个共同属性进行比较等。在少文档干扰设置中,QA 模型有 10 个段落,保证能找到黄金段落;在开放域全维基设置中,模型只给出问题和整个维基百科。模型根据其答案准确性和可解释性进行评估,其中前者被测量为具有完全匹配 (EM) 和 unigram F1 的预测答案和黄金答案之间的重叠,后者关注预测的支持事实句子与人类注释的匹配程度(Supporting Fact EM/F1)。该数据集还报告了一个联合指标,它鼓励系统同时在两项任务上表现良好。 来源:通过迭代查询生成回答复杂的开放域问题
OpenDataLab 收录
VEDAI
用于训练YOLO模型的VEDAI数据集,包含图像和标签,用于目标检测和跟踪。
github 收录
DeepMIMO dataset
DeepMIMO数据集是一个公开的数据集生成框架,用于模拟多种无线通信环境。该数据集特别适用于大规模MIMO和毫米波应用,通过精确的射线追踪数据构建信道模型。数据集包含多个场景,每个场景都有详细的参数设置,如频率、带宽、基站天线配置等。这些参数可以根据研究需求进行调整,以生成符合特定研究目的的数据集。DeepMIMO数据集广泛用于无线通信系统的定位和性能评估研究,特别是在5G及未来通信技术中。
arXiv 收录
