PDBEurope/protein_structure_NER_model_v1.4|生物学数据集|蛋白质结构分析数据集
收藏数据集概述
数据集信息
- 许可证: MIT
- 语言: 英语
- 标签: 生物学, 蛋白质结构, 标记分类
- 配置:
- 配置名称: protein_structure_NER_model_v1.4
- 数据文件:
- 训练集:
annotation_IOB/train.tsv - 开发集:
annotation_IOB/dev.tsv - 测试集:
annotation_IOB/test.tsv
- 训练集:
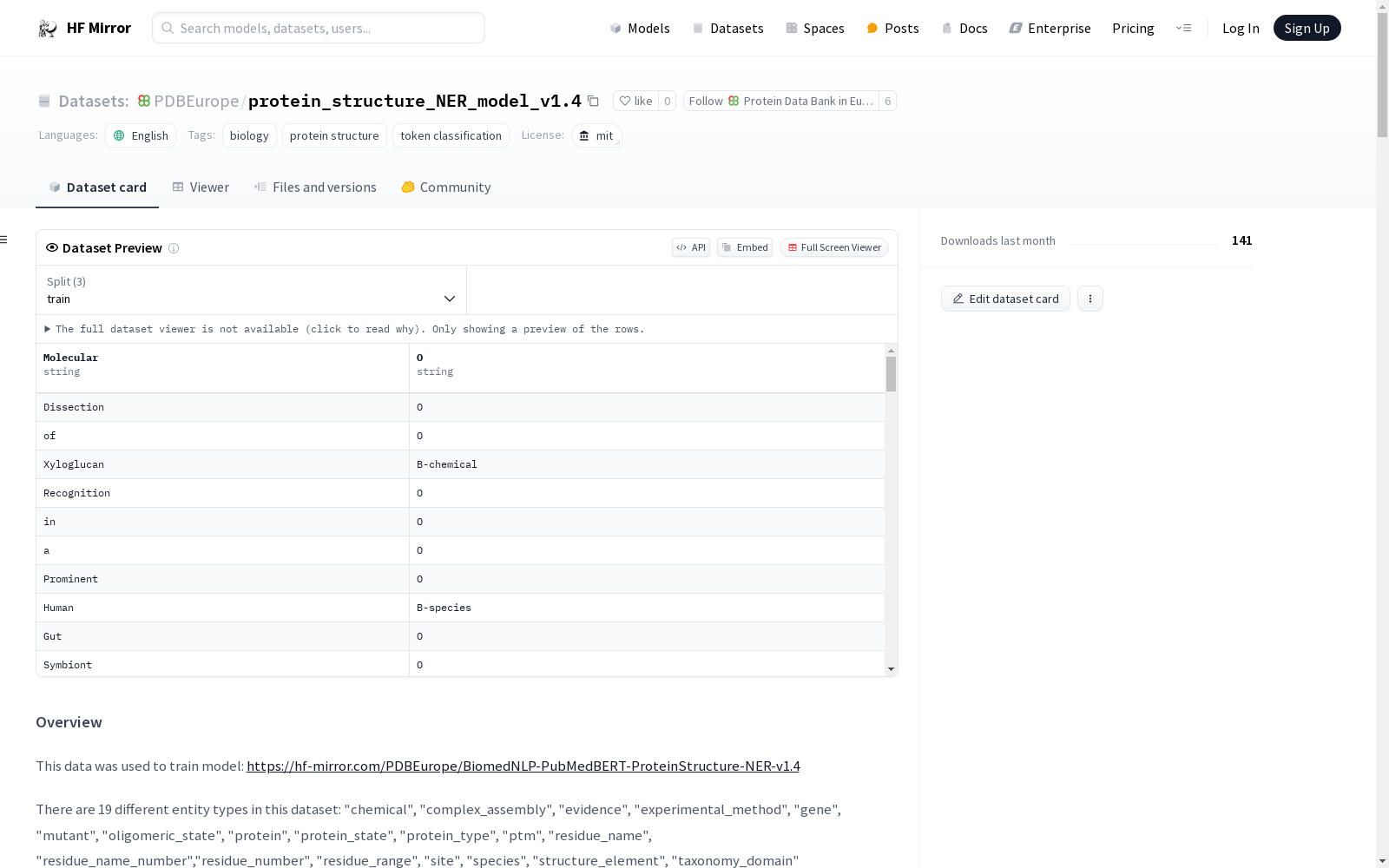
实体类型
数据集中包含19种不同的实体类型:
- chemical
- complex_assembly
- evidence
- experimental_method
- gene
- mutant
- oligomeric_state
- protein
- protein_state
- protein_type
- ptm
- residue_name
- residue_name_number
- residue_number
- residue_range
- site
- species
- structure_element
- taxonomy_domain
数据格式
数据以IOB格式准备,用于训练、开发和测试。此外,还提供了JSON、XML和CSV格式的数据。
注释信息
注释使用免费的注释工具TeamTat进行,文档以BioC XML格式下载,然后转换为IOB、JSON和CSV格式。
注释数量和句子数量
| 文档ID | BioC XML注释数量 | IOB/JSON/CSV注释数量 | 句子数量 |
|---|---|---|---|
| PMC4850273 | 1121 | 1121 | 204 |
| PMC4784909 | 865 | 865 | 204 |
| PMC4850288 | 716 | 708 | 146 |
| PMC4887326 | 933 | 933 | 152 |
| PMC4833862 | 1044 | 1044 | 192 |
| PMC4832331 | 739 | 718 | 134 |
| PMC4852598 | 1229 | 1218 | 250 |
| PMC4786784 | 1549 | 1549 | 232 |
| PMC4848090 | 987 | 985 | 191 |
| PMC4792962 | 1268 | 1268 | 256 |
| PMC4841544 | 1434 | 1433 | 273 |
| PMC4772114 | 825 | 825 | 166 |
| PMC4872110 | 1276 | 1276 | 253 |
| PMC4848761 | 887 | 883 | 252 |
| PMC4919469 | 1628 | 1616 | 336 |
| PMC4880283 | 771 | 771 | 166 |
| PMC4937829 | 625 | 625 | 181 |
| PMC4968113 | 1238 | 1238 | 292 |
| PMC4854314 | 481 | 471 | 139 |
| PMC4871749 | 383 | 383 | 76 |
| 总计 | 19999 | 19930 | 4095 |
数据文件
原始BioC XML文件
- 目录:
raw_BioC_XML - 命名规则:
unique PubMedCentral ID_raw.xml
注释IOB格式
- 目录:
annotation_IOB - 文件:
all.tsv: 所有句子和注释,用于创建模型 "PDBEurope/BiomedNLP-PubMedBERT-ProteinStructure-NER-v1.4",包含4095个句子train.tsv: 训练子集,包含2866个句子dev.tsv: 开发子集,包含614个句子test.tsv: 测试子集,包含615个句子
- 总注释数量: 19930
注释BioC JSON格式
- 目录:
annotated_BioC_JSON - 命名规则:
unique PubMedCentral ID_ann.json - 内容:
sourceid: 唯一PubMedCentral ID的数字部分text: 出版物的完整原始文本denotations: 文本的所有注释列表
注释BioC XML格式
- 目录:
annotated_BioC_XML - 命名规则:
unique PubMedCentral ID_ann.xml - 关键标签:
passage: 包含注释的文本段落offset: 段落偏移量,用于确定注释的起始和结束位置text: 段落的原始文本
注释CSV格式
- 目录:
annotation_CSV - 命名规则:
unique PubMedCentral ID.csv - 列标签:
anno_start: 注释的起始字符位置anno_end: 注释的结束字符位置anno_text: 注释覆盖的文本entity_type: 注释的实体类型sentence: 包含注释的句子文本section: 注释所在的出版物部分
注释JSON格式
- 目录:
annotation_JSON - 文件名:
annotations.json - 内容:
PMC4850273: 唯一PubMedCentral IDannotations: 文档的相关注释句子列表,每个句子包含以下子键:sid: 唯一句子IDsent: 句子文本section: 句子所在的出版物部分ner: 嵌套的注释列表,每个子列表包含起始字符位置、结束字符位置、注释文本和实体类型
MedDialog
MedDialog数据集(中文)包含了医生和患者之间的对话(中文)。它有110万个对话和400万个话语。数据还在不断增长,会有更多的对话加入。原始对话来自好大夫网。
github 收录
中国交通事故深度调查(CIDAS)数据集
交通事故深度调查数据通过采用科学系统方法现场调查中国道路上实际发生交通事故相关的道路环境、道路交通行为、车辆损坏、人员损伤信息,以探究碰撞事故中车损和人伤机理。目前已积累深度调查事故10000余例,单个案例信息包含人、车 、路和环境多维信息组成的3000多个字段。该数据集可作为深入分析中国道路交通事故工况特征,探索事故预防和损伤防护措施的关键数据源,为制定汽车安全法规和标准、完善汽车测评试验规程、
北方大数据交易中心 收录
China Health and Nutrition Survey (CHNS)
China Health and Nutrition Survey(CHNS)是一项由美国北卡罗来纳大学人口中心与中国疾病预防控制中心营养与健康所合作开展的长期开放性队列研究项目,旨在评估国家和地方政府的健康、营养与家庭计划政策对人群健康和营养状况的影响,以及社会经济转型对居民健康行为和健康结果的作用。该调查覆盖中国15个省份和直辖市的约7200户家庭、超过30000名个体,采用多阶段随机抽样方法,收集了家庭、个体以及社区层面的详细数据,包括饮食、健康、经济和社会因素等信息。自2011年起,CHNS不断扩展,新增多个城市和省份,并持续完善纵向数据链接,为研究中国社会经济变化与健康营养的动态关系提供了重要的数据支持。
www.cpc.unc.edu 收录
GME Data
关于2021年GameStop股票活动的数据,包括每日合并的GME短期成交量数据、每日失败交付数据、可借股数、期权链数据以及不同时间框架的开盘/最高/最低/收盘/成交量条形图。
github 收录
FER2013
FER2013数据集是一个广泛用于面部表情识别领域的数据集,包含28,709个训练样本和7,178个测试样本。图像属性为48x48像素,标签包括愤怒、厌恶、恐惧、快乐、悲伤、惊讶和中性。
github 收录
